Os resultados podem abrir o caminho para medicamentos antivirais de amplo espectro contra ZIKV.
Embora inicialmente descrito como uma versão moderada da dengue, o surto do vírus Zika (ZIKV) nas Américas inesperadamente revelou impactos neurológicos importantes como microcefalia fetal ou outros danos cerebrais congênitos quando as mulheres são infectadas durante a gravidez e síndrome de Guillain-Barre (uma doença em que o sistema imunológico do corpo danifica os nervos) em adultos. Pode ser transmitida tanto pelo vetor inseto quanto pelo contato sexual. Este surto tornou-se uma ameaça à saúde global, de epidemiologia complexa e impactos neurológicos devastadores, exigindo, portanto, esforços urgentes para o desenvolvimento de novos medicamentos antivirais eficazes e seguros.
Um dos principais alvos para a descoberta de fármacos é a proteína não-estrutural 5 (NS5) RNA-dependente RNA-polimerase (RdRp) que é codificada nos genomas de todos os vírus contendo RNA sem fase de DNA e tem um papel central na replicação de RNA viral.
Embora os inibidores da polimerase nucleosídica (NPIs) tenham alcançado sucesso clínico no caso de infecções pelo vírus da hepatite C (por exemplo, sofosbovir), eles dependem da ativação por enzimas quinases do hospedeiro e são potencialmente sujeitos a problemas de toxicidade. Deste modo, não-NPIs são ativamente procurados como inibidores de NS5 RdRp de flavivirus (como os vírus da dengue, vírus do Nilo Ocidental, vírus da febre amarela, entre outros), tendo particularmente como alvo o chamado priming loop, que regula a ligação e a polimerização do RNA. Recentemente, vários grupos relataram a descoberta de novos inibidores de RdRp com atividade pan-serótipo contra o vírus da dengue (DENV). Nestes casos, o uso de estruturas cristalográficas de raios X foi fundamental para a identificação de substancias candidatas para o desenvolvimento de fármacos [1, 2].
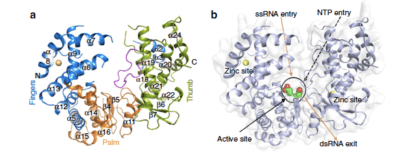
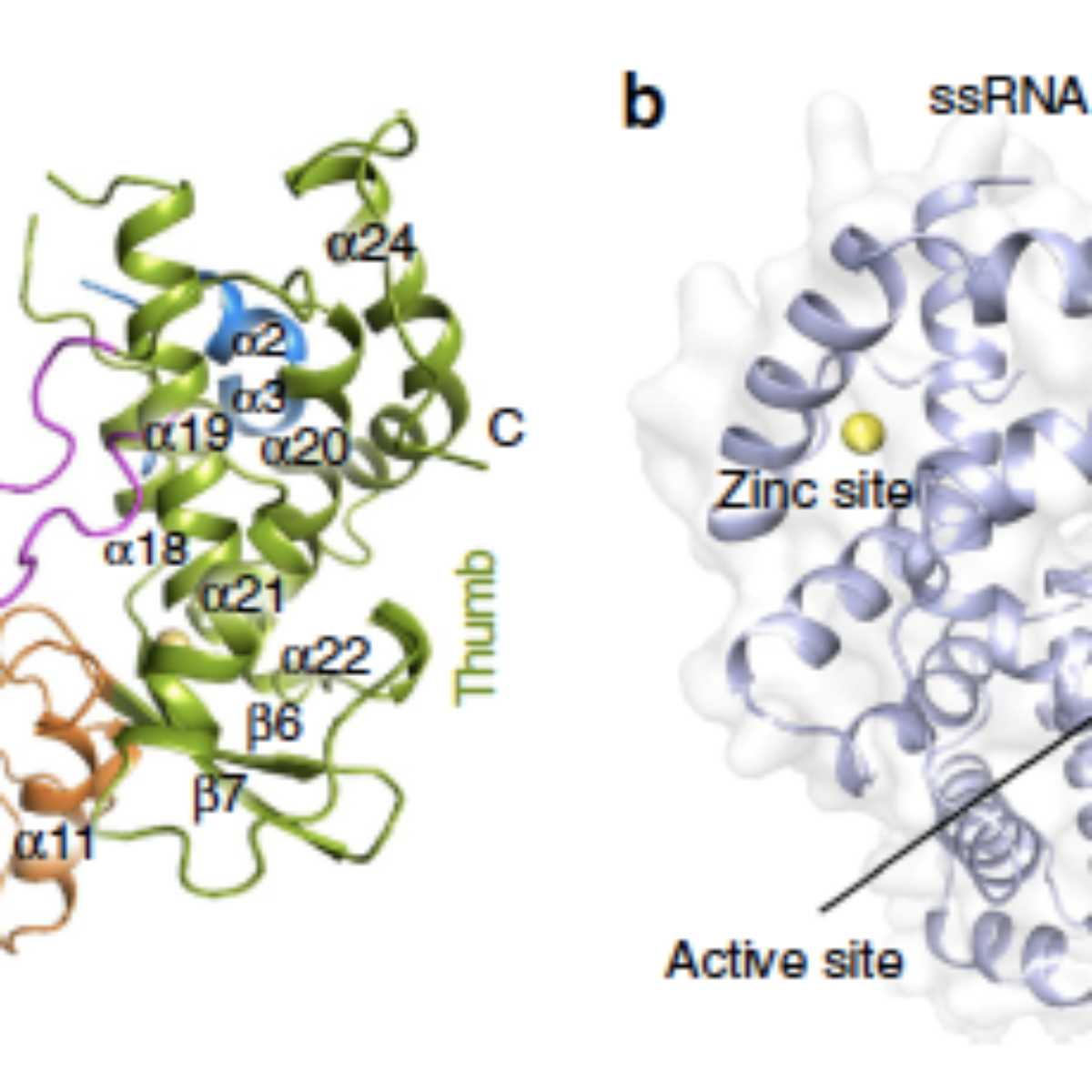
Figura 1. Estrutura cristalina da proteína NS5 RdRp do vírus Zika. (A) Estrutura de RdRp de ZIKV NS5 com domínios de dedos, palma e polegar coloridos em azul, laranja e verde, respectivamente. O priming loop é representado em rosa. (B) Vista da superfície do ZIKV NS5 RdRp, com setas cor de laranja apontando a entrada do molde de RNA de cadeia simples e a região de saída do RNA de cadeia dupla. Aspartatos catalíticos são representados como esferas verdes / vermelhas. A seta tracejada preta mostra o caminho de entrada de NTPs, enquanto a seta preta aponta para a posição do sítio ativo. Em ambas as figuras, os átomos de zinco são representados em amarelo.
Recentemente, Andre S. Godoy e colaboradores [3] determinaram a estrutura cristalina do domínio RdRp NS5 do ZIKV em alta resolução (1,9 Å) e a compararam com as proteínas homólogas do vírus da dengue de diferentes serótipos para identificar locais alvo adequados para a descoberta de fármacos anti-ZIKV e elucidar as suas propriedades estruturais para a ligação de fármacos. A estrutura geral é semelhante a outro homólogo flaviviral, no entanto, com o local alvo do priming loop, que é adequado para NPIs, incluindo uma cavidade com volume menor e uma distribuição de carga local modificada.
Os cristais isolados foram crio-resfriados em nitrogênio líquido e os dados de difração de raios X foram coletados na linha de luz MX2 do Laboratório Nacional de Luz Sincrotron (LNLS) usando um comprimento de onda de 1.4586 Å. Apesar das semelhanças estruturais com DENV, o ZIKV NS5 RdRp exibe diferenças significativas no sítio de ligação do priming loop que pode afetar o design de ligantes.
Essas descobertas podem abrir caminho para a descoberta e o desenvolvimento de fármacos antivirais de amplo espectro contra Flaviviridae, incluindo ZIKV.
Fontes:
[1] Yokokawa, F. et al. Discovery of potent non-nucleoside inhibitors ofdengue viral RNA-dependent RNA polymerase from a fragment hit using structure-based drug design. J. Med. Chem. 59, 3935–3952 (2016). DOI: 10.1021/acs.jmedchem.6b00143
[2] Anusuya, S., Velmurugan, D. & Gromiha, M. M. Identification of dengue viral RNA-dependent RNA polymerase inhibitor using computational fragmentbased approaches and molecular dynamics study. J. Biomol. Struct. Dyn. 34,1512–1532 (2016). DOI: 10.1080/07391102.2015.1081620
[3] Andre S. Godoy, Gustavo M.A. Lima, Ketllyn I.Z. Oliveira, Naiara U. Torres, Fernando V. Maluf, Rafael V.C. Guido & Glaucius Oliva. Crystal structure of Zika virus NS5 RNA-dependent RNA polymerase. Nature Communications 8, 14764, 2017. DOI:10.1038/ncomms14764
Pesquisadores investigam a preservação de partes moles durante fossilização
Reação é um passo importante na transformação de Gás Carbônico em Combustíveis